摘要:一个全新的抗生素世界正等待着感染细菌的病毒。科学家们正在使研究它们变得更容易。
随着传统抗生素对不断进化的病原体不断失去效力,科学家们热衷于使用噬菌体(感染细菌的病毒)完善的杀菌技术。
他们面临的一个主要挑战是难以研究单个噬菌体蛋白,并精确确定病毒如何利用这些工具杀死宿主细菌。劳伦斯伯克利国家实验室的新研究可能有助于加快这一进程。
伯克利实验室生物科学领域的Vivek Mutalik说“我们开发了一种高通量基因筛查方法,可以识别一种称为‘单基因裂解蛋白(single-gene lysis proteins)’的强效噬菌体武器靶向的细菌细胞的部分,随着抗生素耐药性的上升,我们迫切需要抗生素替代品。我们所知道的一些最小的噬菌体编码单基因裂解蛋白(Sgls),也被称为‘蛋白质抗生素(protein antibiotics)’,用于抑制细菌细胞壁生产的关键成分,当这些成分被破坏时,会持续杀死细胞。”

图1 开发了一种高通量基因筛查方法识别单基因裂解蛋白(single-gene lysis proteins)
每一种已知的细菌都至少有一种噬菌体,它们被认为是地球上数量最多的生物实体。事实上,目前地球上估计有1031个噬菌体粒子,相当于每一粒沙子有一万亿个噬菌体。这些噬菌体中的每一个都与它们所选择的宿主菌株一起进化,使它们能够利用改进的生物武器对抗细菌抗性性状。
这种巨大的丰富性、特异性和有效性意味着有很多东西可以研究,理论上我们应该能够使用噬菌体来控制任何有害微生物。噬菌体对非细菌细胞也无害,这是它们作为药物和生物防治工具如此有吸引力的另一个原因。
但当试图将单个噬菌体从环境中分离出来,并确定它针对哪种微生物以及如何靶向时,问题就出现了。科学家们通常无法仅根据基因组序列来评估噬菌体与细菌的战斗,也无法对它们进行实际研究,因为许多细菌无法在实验室中培养——即使可以,也存在一个内在的矛盾,即需要提前知道培养哪些细菌,以便研究感染和杀死它们的噬菌体。
为了避开这些障碍并识别Sgls的细胞靶标,Mutalik和他的同事使用了该团队之前发明的一种称为双条形码散弹枪表达库测序(Dub-seq,生物通注)的技术。Dub-seq允许科学家使用编码的DNA片段库来研究未知基因的功能,并且可以应用于包含许多生物DNA的复杂环境样本——不需要培养。
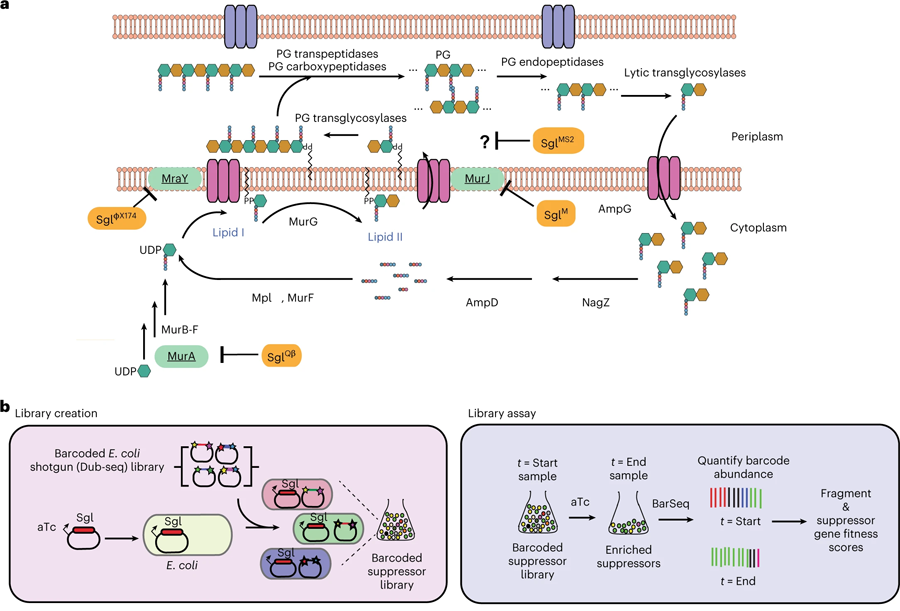
图2 全基因组筛选用于鉴定噬菌体编码单基因裂解系统的宿主抑制因子
在这项研究中,作者使用了来自6种噬菌体的6种Sgls来感染不同的细菌,并确定了每种Sgl攻击的细菌细胞壁的部分或支持分子。他们与德克萨斯A& M大学的科学家合作,对一种Sgl的功能进行了详细的描述。
这项工作表明,Sgl蛋白靶向的细胞壁构建途径在细菌进化史的早期就出现了,并且仍然被几乎所有的细菌(包括致病菌)使用。由于Sgl蛋白攻击这些基本和普遍存在的目标,它们可以杀死噬菌体目标菌株以外的细菌,这证实了它们作为抗生素的巨大潜力。
“在摧毁细菌方面,噬菌体是非凡的创新者。我们真的很高兴能发现新的细菌病原体靶向机制,可以用于治疗,”文章一作Benjamin Adler说,他是加州大学伯克利分校Jennifer Doudna实验室的博士后。
现在,该团队已经评估了Dub-seq方法来解决这个问题,他们可以将其应用于数千个单基因裂解产生噬菌体的环境样本中,该团队从海洋、土壤,甚至人类肠道中收集到的样本。下一个突破性医学的灵感可能就在那里,等待着。
参考资料:
[1] Benjamin A. Adler, Karthik Chamakura, Heloise Carion, Jonathan Krog, Adam M. Deutschbauer, Ry Young, Vivek K. Mutalik, Adam P. Arkin. Multicopy suppressor screens reveal convergent evolution of single-gene lysis proteins. Nature Chemical Biology, 2023; DOI: 10.1038/s41589-023-01269-7
摘要:一个全新的抗生素世界正等待着感染细菌的病毒。科学家们正在使研究它们变得更容易。
随着传统抗生素对不断进化的病原体不断失去效力,科学家们热衷于使用噬菌体(感染细菌的病毒)完善的杀菌技术。
他们面临的一个主要挑战是难以研究单个噬菌体蛋白,并精确确定病毒如何利用这些工具杀死宿主细菌。劳伦斯伯克利国家实验室的新研究可能有助于加快这一进程。
伯克利实验室生物科学领域的Vivek Mutalik说“我们开发了一种高通量基因筛查方法,可以识别一种称为‘单基因裂解蛋白(single-gene lysis proteins)’的强效噬菌体武器靶向的细菌细胞的部分,随着抗生素耐药性的上升,我们迫切需要抗生素替代品。我们所知道的一些最小的噬菌体编码单基因裂解蛋白(Sgls),也被称为‘蛋白质抗生素(protein antibiotics)’,用于抑制细菌细胞壁生产的关键成分,当这些成分被破坏时,会持续杀死细胞。”

图1 开发了一种高通量基因筛查方法识别单基因裂解蛋白(single-gene lysis proteins)
每一种已知的细菌都至少有一种噬菌体,它们被认为是地球上数量最多的生物实体。事实上,目前地球上估计有1031个噬菌体粒子,相当于每一粒沙子有一万亿个噬菌体。这些噬菌体中的每一个都与它们所选择的宿主菌株一起进化,使它们能够利用改进的生物武器对抗细菌抗性性状。
这种巨大的丰富性、特异性和有效性意味着有很多东西可以研究,理论上我们应该能够使用噬菌体来控制任何有害微生物。噬菌体对非细菌细胞也无害,这是它们作为药物和生物防治工具如此有吸引力的另一个原因。
但当试图将单个噬菌体从环境中分离出来,并确定它针对哪种微生物以及如何靶向时,问题就出现了。科学家们通常无法仅根据基因组序列来评估噬菌体与细菌的战斗,也无法对它们进行实际研究,因为许多细菌无法在实验室中培养——即使可以,也存在一个内在的矛盾,即需要提前知道培养哪些细菌,以便研究感染和杀死它们的噬菌体。
为了避开这些障碍并识别Sgls的细胞靶标,Mutalik和他的同事使用了该团队之前发明的一种称为双条形码散弹枪表达库测序(Dub-seq,生物通注)的技术。Dub-seq允许科学家使用编码的DNA片段库来研究未知基因的功能,并且可以应用于包含许多生物DNA的复杂环境样本——不需要培养。

图2 全基因组筛选用于鉴定噬菌体编码单基因裂解系统的宿主抑制因子
在这项研究中,作者使用了来自6种噬菌体的6种Sgls来感染不同的细菌,并确定了每种Sgl攻击的细菌细胞壁的部分或支持分子。他们与德克萨斯A& M大学的科学家合作,对一种Sgl的功能进行了详细的描述。
这项工作表明,Sgl蛋白靶向的细胞壁构建途径在细菌进化史的早期就出现了,并且仍然被几乎所有的细菌(包括致病菌)使用。由于Sgl蛋白攻击这些基本和普遍存在的目标,它们可以杀死噬菌体目标菌株以外的细菌,这证实了它们作为抗生素的巨大潜力。
“在摧毁细菌方面,噬菌体是非凡的创新者。我们真的很高兴能发现新的细菌病原体靶向机制,可以用于治疗,”文章一作Benjamin Adler说,他是加州大学伯克利分校Jennifer Doudna实验室的博士后。
现在,该团队已经评估了Dub-seq方法来解决这个问题,他们可以将其应用于数千个单基因裂解产生噬菌体的环境样本中,该团队从海洋、土壤,甚至人类肠道中收集到的样本。下一个突破性医学的灵感可能就在那里,等待着。
参考资料:
[1] Benjamin A. Adler, Karthik Chamakura, Heloise Carion, Jonathan Krog, Adam M. Deutschbauer, Ry Young, Vivek K. Mutalik, Adam P. Arkin. Multicopy suppressor screens reveal convergent evolution of single-gene lysis proteins. Nature Chemical Biology, 2023; DOI: 10.1038/s41589-023-01269-7



